Oligonukleotydy – krótkie cząsteczki DNA lub RNA, które mają szeroki zakres zastosowania, między innymi w farmacji, genetyce czy kryminalistyce. Są one powszechnie wytwarzane w laboratorium przez syntezę chemiczną na podłożu stałym i mogą być otrzymywane jako cząsteczki jednoniciowe o dowolnie określonej sekwencji, co sprawia, że są niezbędne do syntezy sztucznych genów, reakcji łańcuchowej polimerazy (PCR), sekwencjonowania DNA, klonowania molekularnego i sond molekularnych. W przyrodzie oligonukleotydy zazwyczaj występują jako małe cząsteczki RNA, które biorą udział w regulacji ekspresji genu poprzez mechanizm interferencji RNA (miRNA i siRNA) lub w innych procesach regulacyjnych (np. piRNA lub scRNA), lub są produktami przejściowymi degradacji większych cząsteczek kwasu nukleinowego.
Sekwencję oligonukleotydów określa liczba i rodzaj nukleotydów, które tworzą całą cząsteczkę. Długość oligonukleotydu jest zwykle oznaczana przez "-mer" (z greckiego meros, "część"). Przykładowo, oligonukleotyd składający się z sześciu nukleotydów jest heksamerem, podczas gdy złożony z 25 nukleotydów to pentakozamer, zwykle nazywany "25-merem". Oligonukleotydy łatwo wiążą się, w sposób specyficzny dla danej sekwencji, z odpowiednimi komplementarnymi oligonukleotydami DNA lub RNA, tworząc dupleksy lub, rzadziej, hybrydy wyższego rzędu.
Synteza
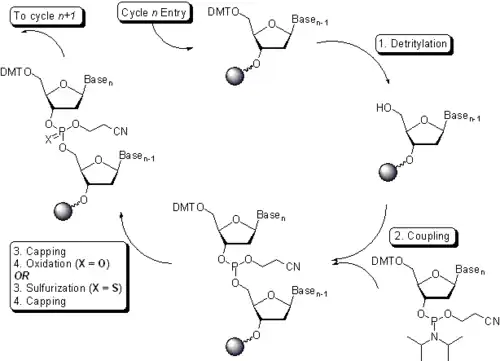
Syntetyczne oligonukleotydy mogą mieć budowę identyczną z naturalną lub być modyfikowane, w części fosforanowej, w pierścieniu rybozy/deoksyrybozy lub w części zasady azotowej, np. w celu uzyskania różnych efektów farmakologicznych. Modyfikacje te nadają oligonukleotydom nowe właściwości i są kluczowym elementem wykorzystania ich w terapii, np. antysensowej, jako siRNA, aptamery i in.[1][2][3]
Oligonukleotydy są syntetyzowane chemicznie zazwyczaj zautomatyzowaną metodą amidofosforynową na podłożu stałym. Synteza przebiega w kierunku 3' do 5' i polega na stopniowym dołączaniu kolejnych reszt nukleotydowych do rosnącego łańcucha. Każdy cykl obejmuje cztery reakcje chemiczne: odblokowanie grupy 5'-hydroksylowej, kondensację (dołączenie kolejnej jednostki nukleotydowej), tzw. kapowanie (trwałe zablokowanie nieprzereagowanych grup 5'-OH) i utlenianie fosforynu do fosforanu. Po każdej z nich podłoże jest przemywane rozpuszczalnikiem w celu usunięcia produktów ubocznych i nieprzereagowanych substratów[4][5]. Wydajność końcowa całej syntezy Wk wynosi[4]:
- Wk = Wn × 100%
- gdzie n – liczba cykli syntetycznych, Wn – średnia wydajność jednego cyklu wyrażona jako ułamek molowy
Aby uzyskać pożądany oligonukleotyd konieczne jest osiągnięcie wydajności bliskiej 100% dla każdej reakcji podczas syntezy[4]. Typowa długość syntetycznych oligonukleotydowe jest rzędu 20 nukleotydów[6], a ich maksymalna długość może wynosić do 300 nukleotydów[7]. Po syntezie usuwane są grupy blokujące, a produkt odłączany jest od podłoża, co uzyskuje się np. ogrzewając podłoże z oligonukleotydem w stężonym roztworze amoniaku. Do izolowania oraz oczyszczania uzyskanych łańcuchów oligonukleotydowych stosuje się HPLC lub elektroforezę[5].
Oligonukleotydy można też otrzymać na drodze hydrolizy (chemicznej lub enzymatycznej) kwasów nukleinowych oraz syntezy enzymatycznej[8].
Modyfikacje oligonukleotydów
Tworzenie stabilnych chemicznie krótkich oligonukleotydów było najwcześniejszym wyzwaniem w rozwijaniu terapii antysensowej. Oligonukleotydy o budowie naturalnej są szybko rozkładane przez nukleazy, występujęce w każdym typie komórek, a ponadto, ze względu na ujemny ładunek reszt fosforanowych, słabo przenikają przez błony komórkowe. Z tego względu, aby wykorzystać oligonukleotydy do celów terapeutycznych, konieczne jest wprowadzenie modyfikacji, które zwiększają ich stabilność i lipofilowość[9][10].
Modyfikacje grupy fosforanowej
Najczęściej stosowaną modyfikacją grupy fosforanowej oligonukleotydów jest zamiana jednego z niewiążących atomów tlenu na atom siarki, a zatem zastąpienie naturalnej reszty fosforanowej resztą tiofosforanową. Jeden z niemostkowych atomów tlenu zostaje wówczas zastąpiony atomem siarki, dzięki czemu odporność na degradację enzymatyczną wzrasta o kilka rzędów wielkości. Innymi modyfikkacjami wiązania internukleotydowego są metylofosfoniany (P−CH
3), boranofosfoniany (P−BH−
3), amidofosforany (P−NR
2) i in.[10]
Modyfikacje w części cukrowej
Przykładowe modyfikacje w pozycji 2', to grupy 2'-O-metylowa i 2'-O-metoksyetylowa[10].
Oligonukleotydy antysensowe
Oligonukleotydy antysensowe (ASO, z ang. antisense oligonucleotides) są pojedynczymi nićmi DNA lub RNA, które są komplementarne do wybranej sekwencji mRNA. Po wytworzeniu dupleksów typu RNA-DNA, nić mRNA jest degradowana przez RNazę H, wstrzymując syntezę niepożądanego białka[11].
Morfolino oligonukleotydy są grupa antysensowych oligonukleotydów, które są stosowane w biologii molekularnej do modyfikacji ekspresji genów. Ich struktura zawiera zasady DNA przyłączone do szkieletu pierścieni metylenomorfolinowych połączonych grupami fosforodiamidowymi. Zatwierdzone przez FDA leki golodirsen i eteplirsen, które są przeznaczone do leczenia DMD zawierają w swojej strukturze morfolino oligonukleotydy[12].
Techniki analityczne
Oligonukleotydy mogą być analizowane za pomocą:
- wysokosprawnej chromatografii cieczowej z fazą odwrócona (RP-HPLC)[5][13]
- elektroforezy w żelu poliakrylamidowym (PAGE, z ang. poluacrylamide gel electrophoresis)[5]
- spektrometrii mas – stosuje się głównie dwie techniki jonizacji: desorpcja laserowa z udziałem matrycy (ang. matrix assisted laser desorption ionisation, MALDI) oraz elektrorozpylanie (ang. electrospray ionization, ESI)[14].
- mikromacierzy DNA, pozwalających na badanie ekspresji genów, jak również w innych badaniach biologicznych, medycznych i chemicznych[15].
Przypisy
- ↑ Chisato Terada i inni, Chemistry of Therapeutic Oligonucleotides That Drives Interactions with Biomolecules, „Pharmaceutics”, 14 (12), 2022, s. 2647, DOI: 10.3390/pharmaceutics14122647, PMID: 36559141, PMCID: PMC9781680 [dostęp 2023-03-23] (ang.).
- ↑ Bingchuan Wei, Alexandre Goyon, Kelly Zhang, Analysis of therapeutic nucleic acids by capillary electrophoresis, „Journal of Pharmaceutical and Biomedical Analysis”, 219, 2022, s. 114928, DOI: 10.1016/j.jpba.2022.114928 [dostęp 2023-03-23] (ang.).
- ↑ Stefan Jurga, Volker A. Erdmann, Jan Barciszewski (red.), Modified Nucleic Acids in Biology and Medicine, Cham, Szwajcarja): Springer International Publishing, 2016, DOI: 10.1007/978-3-319-34175-0, ISBN 978-3-319-34173-6, OCLC 955018659 (ang.).
- 1 2 3 Izabela Burzyńska-Pędziwiatr, Lucyna Woźniak, Synteza chemiczna oligonukleotydów na fazie stałej. Możliwości i ograniczenia nośników stałych, „Wiadomości Chemiczne”, 62 (7-8), 2008, s. 571-594 [dostęp 2023-03-23].
- 1 2 3 4 Serge L. Beaucage, Oligodeoxyribonucleotides Synthesis. Phosphoramidite Approach, [w:] Sudhir Agrawal (red.), Protocols for oligonucleotides and analogs. Synthesis and properties, t. 20, New Jersey: Humana Press, 1993 (Methods in Molecular Biology, vol 20), s. 33–62, DOI: 10.1385/0-89603-281-7:33, ISBN 978-0-89603-281-1, OCLC 55638684 (ang.).
- ↑ Jimin Yang i inni, Solid-Phase Synthesis of Phosphorothioate Oligonucleotides Using Sulfurization Byproducts for in Situ Capping, „Journal of Organic Chemistry”, 83 (19), 2018, s. 11577–11585, DOI: 10.1021/acs.joc.8b01553 [dostęp 2023-03-23] (ang.).
- ↑ Dhananjani N.A.M. Eriyagama i inni, Parallel, Large-Scale, and Long Synthetic Oligodeoxynucleotide Purification Using the Catching Full-Length Sequence by Polymerization Technique, „Organic Process Research & Development”, 22 (9), 2018, s. 1282–1288, DOI: 10.1021/acs.oprd.8b00209, PMID: 30906183, PMCID: PMC6428204 [dostęp 2023-03-23] (ang.).
- ↑ Andrzej Okruszek, Metody syntezy modyfikowanych oligonukleotydów i ich analogów potencjalnych inhibitorów ekspresji genów w strategii antysensowej, „Biotechnologia”, 4/1994, s. 16-39, ISSN 0860-7796 [dostęp 2023-03-23].
- ↑ Eugen Uhlmann, Anusch Peyman, Antisense oligonucleotides: a new therapeutic principle, „Chemical Reviews”, 90 (4), 1990, s. 543–584, DOI: 10.1021/cr00102a001 [dostęp 2023-03-23] (ang.).
- 1 2 3 John Goodchild, Conjugates of oligonucleotides and modified oligonucleotides: a review of their synthesis and properties, „Bioconjugate Chemistry”, 1 (3), 1990, s. 165–187, DOI: 10.1021/bc00003a001 [dostęp 2023-03-23] (ang.).
- ↑ Wojciech J. Stec, Strategia antysensowych oligonukleotydów, „Biotechnologia”, 4/1994, s. 5-15, ISSN 0860-7796 [dostęp 2023-03-23].
- ↑ David R. Corey, John M. Abrams, Morpholino antisense oligonucleotides: tools for investigating vertebrate development, „Genome Biology”, 2 (5), 2001, reviews1015.1–1015.3, DOI: 10.1186/gb-2001-2-5-reviews1015, PMID: 11387041, PMCID: PMC138935 [dostęp 2023-03-23].
- ↑ Michael Swartz, HPLC detectors: a brief review, „Journal of Liquid Chromatography & Related Technologies”, 33 (9-12), 2010, s. 1130–1150, DOI: 10.1080/10826076.2010.484356 [dostęp 2023-03-23] (ang.).
- ↑ Anas El-Aneed, Aljandro Cohen, Joseph Banoub, Mass Spectrometry, Review of the Basics: Electrospray, MALDI, and Commonly Used Mass Analyzers, „Applied Spectroscopy Reviews”, 44 (3), 2009, s. 210–230, DOI: 10.1080/05704920902717872 [dostęp 2023-03-23] (ang.).
- ↑ Agnieszka Kisiel i inni, Mikromacierze DNA, „Kosmos”, 53 (3–4), 2004, s. 295–303 [dostęp 2023-03-23].